Genetisk inomartsvariation
Övervakning av den marina miljön ger underlag för att bedöma och följa utvecklingen av miljöns tillstånd och de miljöförändringar som uppstår till följd av mänskliga aktiviteter. Insamlade data kommer till användning i förvaltningen av havet och våra marina ekosystem.

Lokalt anpassade bestånd av sill i Östersjön. Foto: Leif Andersson
Sill (Clupea harengus) har ett stort antal lokalt anpassade bestånd. Överst, vårlekande strömming (till vänster) och höstlekande strömming (till höger). Underst, slåttersill från Gävlebukten som är betydligt större och bland annat är fiskätande till skillnad från strömming som framförallt äter djurplankton.
Övergripande information
De flesta individer är genetiskt olika och genetisk mångfald är grunden för all biologisk mångfald. Genetisk variation inom populationer och arter är en förutsättning för naturligt urval och ärftliga förändringar som leder till anpassning.
Övervakningen av arters genetiska variation har flera syften
- Att identifiera biologiska populationer av arter som i möjligaste mån bör representera förvaltningsenheter (till exempel bestånd inom arter utsatta för fiske).
- Att bedöma vilka populationer som hotas av genetisk utarmning och risk för utrotning.
- Att identifiera populationer med särskilt värdefull genetisk variation, till exempel populationer anpassade till varmare vatten än andra populationer inom arten.
- Att följa genetiska förändringar över tid som kan inverka på populationernas produktivitet, motståndskraft (resiliens) och ekosystemfunktioner.
- Att vid restaureringar av till exempel bestånd av fisk eller sjögräs bedöma lämpliga givarpopulationer.
Genetisk variation i egenskaper som påverkar överlevnad och reproduktion gör det möjligt för populationer av arter att anpassa sig när miljön förändras. "Slumrande" genetisk variation som är obrukbar i nuvarande livsmiljö kan bli livsviktig efter en miljöförändring och man kan därför inte alltid avgöra vilka genvarianter som blir viktiga i en framtida miljö.
Av resursskäl prioriteras idag särskilt viktigt arter, såsom kommersiellt fiskade arter (exempelvis lax, sill, torsk), biotopbildande arter (exempelvis blåstång, ålgräs).
Havs- och vattenmyndigheten
Nationell akvatisk övervakning.
Programmet som helhet startade 2020 (efter en pilotstart under 2017-2020). Startår för enskilda arter (nedan) varierar dock, och i flera fall har prover insamlats under flera säsonger, eller analyserna har kompletterats med material tidigare insamlat för forskningsändamål.
- Lax 2020
- Öring 2017
- Röding 2022
- Torsk 2020
- Sill 2020
- Ålgräs 2020
- Blåstång 2017
- Flodpärlmussla 2025
Vad Sverige övervakar styrs bland annat av olika EU-lagstiftningar som ställer samma krav på alla EU:s medlemsstater (se Vad styr vad som ska övervakas och hur används data?).
Beskrivning av övervakning
Genetisk data extraherade från DNA ur vävnad från ett representativt antal individer per art samlas in för fem arter av fisk (lax, öring, röding, torsk och sill), två habitatbildande arter (ålgräs och blåstång) samt en mollusk (flodpärlmussla). För varje individ registreras metadata (lokal, tidpunkt, och för flera arter även fenotypiska data).
Lax (Salmo salar)
Genetisk data
SNP-genotyper omfattande mellan knappt 100 och upp till omkring 4000 loci per individ, beroende på om det rör sig om befintliga data från tidigare forskningsprojekt eller nya data genererade inom HaV:s genetiska övervakning.
Analyserade genetiska parametrar
- Genetisk variation i tid och rum (heterozygositet, antal anlagsvarianter, FST)
- inom och mellan lokala populationer (älvbestånd eller delbestånd inom samma älv) samt
- genetiskt baserade skattningar av effektiva populationsstorlekar.
Metadata
- Lokal och datum,
- Individens storlek (längd samt eventuell vikt),
- Kön (endast i vissa fall)
Öring (Salmo trutta)
Genetiska data
SNP-genotyper omfattande cirka 100-3000 loci per individ, helgenomsekvensering av poolade individer respektive individuell sekvensering.
Analyserade genetiska parametrar
- Genetisk variation i tid och rum (heterozygositet, antal anlagsvarianter, nukleotiddiversitet, inavelsgrad, FST,)
- inom och mellan lokala populationer (vattensystem eller delbestånd inom vattensystem) samt
- genetiskt baserade skattningar av effektiva populationsstorlekar.
Metadata
- Lokal och datum,
- Individens storlek (längd, vikt),
- Kön (för vuxen fisk)
- Reproduktivt stadium
- I vissa fall: ålder från otolitanalyser
Röding (Salvelinus alpinus)
Genetiska data
Helgenomsekvensering av individier
Analyserade genetiska parametrar
- Genetisk variation i tid och rum (heterozygositet, antal anlagsvarianter, nukleotiddiversitet, inavelsgrad, FST)
- inom och mellan lokala populationer (vattensystem eller delbestånd inom vattensystem) samt
- genetiskt baserade skattningar av effektiva populationsstorlekar.
Metadata
- Lokal och datum,
- Individens storlek (längd, vikt),
- Kön
- Reproduktivt stadium (vissa prover)
Torsk (Gadus morhua)
Genetiska data
SNP genotyper för 4000 loci för varje individ
Analyserade genetiska parametrar
- Effektiv populationsstorlek (Ne)
- Alleldiversitet ("allelic richness" AR)
- PCA baserat på SNP genotyper - grupperar individer med genetiska likheter
- Genetisk populationsdiversitet, Fst
- Observerad heterozygotigrad, Ho
- Karyotyp av inversion på kromosomerna 2, 7 och 12
Metadata
- Lokal och datum
- Individens storlek (längd och vikt)
- Kön (ej juvenila individer)
Sill (Clupea harengus)
Genetiska data
SNP-genotyper för 4890 loci för varje individ
Analyserade genetiska parametrar
- Basal kartläggning av populationsstruktur
- genetisk variation i tid och rum (heterozygositet, antal anlagsvarianter, inavelsgrad, FST
- inom och mellan genetiska grupperingar) samt
- genetiskt baserade skattningar av effektiva populationsstorlekar.
Metadata
- Lokal och datum,
- Individens storlek (längd, vikt),
- Kön
- Reproduktivt stadium
Ålgräs (Zostera marina)
Genetiska data
SNP genotyper för 2138 loci för varje individ
Analyserade genetiska parametrar
- Observerad heterozygositet (Ho) - beskriver genetisk variation inom en population
- Andel kloner "clonal richness, R" - beskriver diversiteten av kloner på en lokal
- Inavels index (Fis) - negativa värden indikerar graden av asexuell fortplantning
- PCA baserat på SNP genotyper - grupperar individer m.a.p. genetiska likheter
Metadata
Lokal och ålgräsets utbredning på lokalen
- Vattendjup
- Medelvärden på vattentemperatur och salthalt från oceanografisk modellering
- Förekomst av påväxtalger (filamentösa alger eller kiselalger) eller angrepp av parasiten Labyrinthula zosterae
Blåstång (Fucus vesiculosus)
Genetiska data
SNP genotyper för 15409 loci för varje individ
Analyserade genetiska parametrar
- Observerad heterozygositet (Ho) - beskriver genetisk variation inom en population
- Andel kloner "clonal richness, R" - beskriver diversiteten av kloner på en lokal
- Inavels index (Fis) - negativa värden indikerar graden av asexuell fortplantning
- PCA baserat på SNP genotyper - grupperar individer m.a.p. genetiska likheter
- Genetisk populationsdiversitet, Fst - grupperar population m.a.p. genetisk likhet
- Släktskapsanalys, NJ ("neighbour joining tree")
Metadata
- Lokal
- Vattendjup
- Medelvärden på vattentemperatur och salthalt från oceanografisk modellering
- Foto med morfologiska karaktärer (thallusbredd, grentäthet, krusning, adventivgrenar)
Flodpärlmussla (Margaritifera margaritifera)
Genetiska data
Mikrosatellitdata (15 loci för varje individ)
Analyserade genetiska parametrar
- Genetisk variation i tid och rum (heterozygositet, antal anlagsvarianter, FST
- inom och mellan lokala populationer.
Metadata
- Lokal och datum,
- Individens storlek (längd)
- Information om värdart (öring eller lax), om sådan kunskap finns
Atlantlax (Salmo salar)
Löpande genetisk övervakning ungefär vart 6:e år i vattendrag runt hela kusten och i Vänern (se karta).
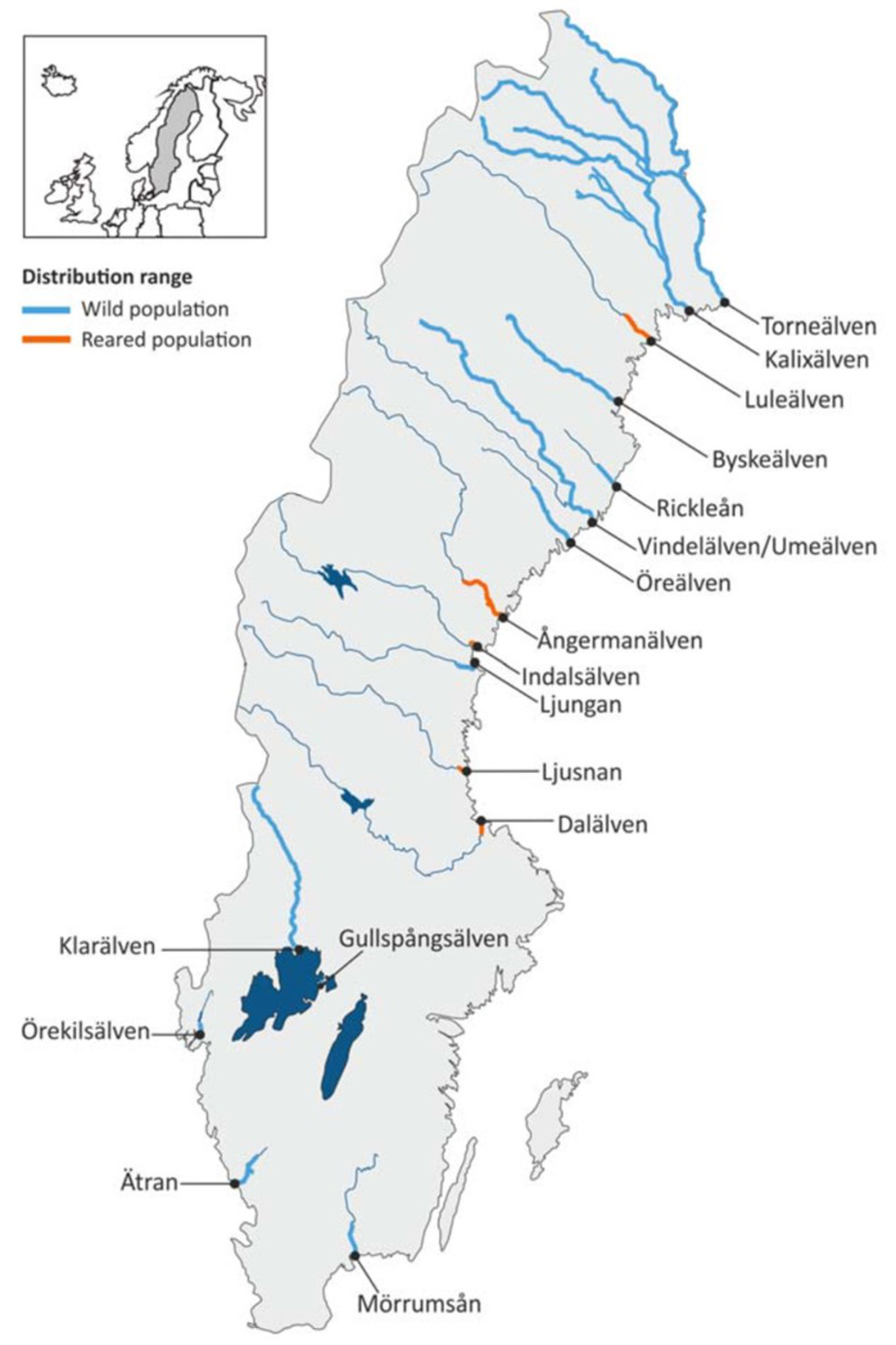
Karta som visar de 17 vattendrag med lax som [hittills] ingått i den genetiska övervakningen. Tjockare linjer markerar nutida utbredningsområden för idag vilda (blå) respektive odlade (röda) populationer.
Öring (Salmo trutta)
Löpande genetisk övervakning ungefär vart 6:e år. Övervakningsprogrammet är under uppbyggnad och omfattar i dagsläget fjällsjööring från Jämtlands län och havsöring från Gotland.
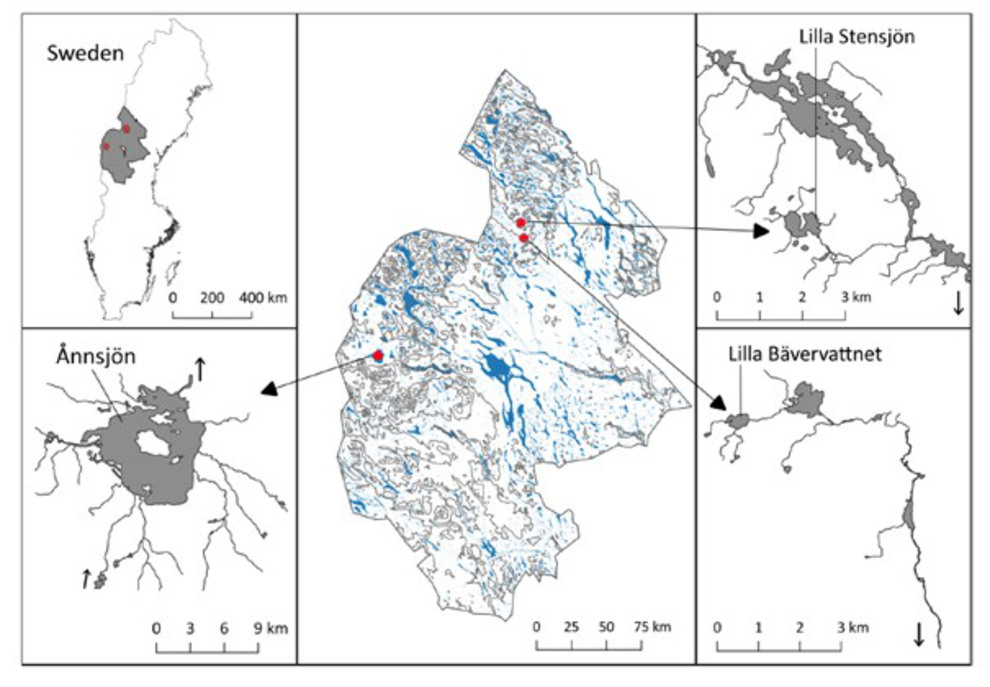
Karta som visar övervakning av genetisk mångfald i skyddade områden i Jämtland.
Torsk (Gadus morhua)
Löpande insamling av genetiska prover sker årligen genom SLU:s provfiske. Hittills har tre olika populationer identifierade med stora genetiska skillnader: östrabeståndet i Östersjön ("EBC"), beståndet i västra Östersjön, Öresund, Kattegatt och Bohusläns fjordar ("coastal"), samt Nordsjöbeståndet ("offshore").
Forskning med flera genetiska markörer (helgenomsekvensering) pågår för att utröna om det finns ett lokalt bestånd runt Åland-Gävlebukten, och om fjordarna i Bohuslän har, eller har haft lokala fjordbestånd.
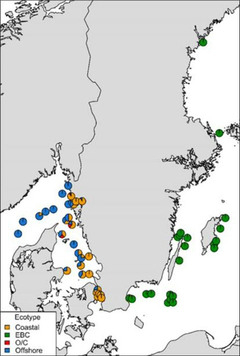
Karta som visar den övervakning som sker avseende torsk. Med ca 4000 genetiska markörer framträder tre olika bestånd, Östersjöns östra bestånd (grön), Öresund/Kattegatt och Bohusläns fjordar (orange) och Nordsjöbeståndet som framförallt leker väster om Danmark men även i utsjön i Skagerrak (blå). Som framgår av kartan överlappar utbredningen av Nordsjöbestånden och Kattegattbeståndet i Kattegatt.
Sill (Clupea harengus)
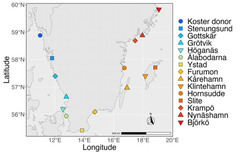
En storskalig kartläggning av sillens (strömmingens) genetiska struktur har genomförts längs östersjökusten som ett första steg i övervakningen. Tre genetiska huvudgrupperingar noteras men genetiskt särpräglade bestånd finns även inom dessa huvudgrupper. Temporal undersökningar pågår på enskilda positioner längs kuststräckan
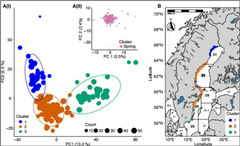
Genetiska kluster för vårlekande sill/strömming i östersjön. Tre genetiska huvudkluster identifierade med SNP-chippet som inkluderar både selekterade och neutrala loci (delfigur (AI)), samt ett enda kluster för vårlekare som identifieras om man bara använder neutrala markörer (A(II)).
Röding (Salvelinus alpinus)
Rödingen har initialt övervakats i tre fjällsjöar i Jämtland för vilka prover funnits tillgängliga från 1970-tal sam 2010-tal. I en av sjöarna har öring inplanterats vid ett tillfälle efter första övervakningspunkten och en avsikt med den initiala övervakningen var att undersöka om effekter av sådan introduktion kan upptäckas jämfört med ”ostörda sjöar” Selektiva skillnader och Beståndet i den påverkade sjön visade större selektiva förändringar över tid.
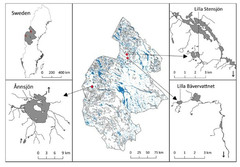
Figur 5. Karta som visar övervakning av genetisk mångfald i tre fjällsjöar i Jämtland.
Ålgräs (Zostera marina)
Löpande genetisk övervakning ungefär vart 6:e år i hela artens svenska utbredningsområde. Antal och plats för provtagning kan komma att modifieras om ny forskning visar behov därav.
Karta som visar övervakning av genetisk mångfald hos ålgräs hela Sveriges utbredningsområde.
Blåstång (Fucus vesiculosus)
Löpande genetisk övervakning ungefär vart 6:e år i hela artens svenska utbredningsområde. Antal och plats för provtagning kan komma att modifieras om ny forskning visar behov därav.
Flodpärlmussla (Margaritifera margaritifera)
Löpande genetisk övervakning cirka vart 6 år i hela artens svenska utbredningsområde.
Genetisk inomartsvariation övervakas genom insamling av vävnad från ett representativt antal individer per population, varefter den genetiska sammansättningen fastställs för varje individ. De primära mätdata som genereras används för att beräkna olika indikatorer som representerar populationers och arters genetiska tillstånd
DNA utvinns genom extraktionsmetoder som i detaljer kan vara specifika för respektive art. (Redovisas i övervakningsmanualerna.) De olika typer av sekvensering av DNA som används redovisas nedan per art. Strävan är att för alla de arter som ingår i genetisk övervakning utveckla en s.k. SNP-panel. Ett identifierat set av några tusentals SNPs (single nucleotide polymorphisms) som fungerar som markörer på de lokala bestånd som identifierats. För fiskarna som ingår i övervakningsprogrammet (alla utom röding) finns idag SNP-paneler samlade på ett så kallatSNP-chip.
För ålgräs och blåstång kommer en SNP-panel att kunna utvecklas från insamlade genetiska data. För flodpärlmussla används så kallade mikrosatelliter. Denna typ av högvariabla DNA-markörer, som har använts under lång tid och täcker en mindre del av arvsmassan än SNP-paneler, har valts för att möjliggöra samkörning och jämförbara analyser av nyinsamlade populationsdata från Sverige med omfattande befintliga data från Norge.
Utifrån de primära mätdata som genereras av den genetiska analysen beräknas tre genetiska tillståndsvärden; effektiv populationsstorlek (Ne), genetisk mångfald inom populationer (ΔH) och genetisk mångfald mellan populationer (ΔFST). Dessa tillståndsvärden är baserade på centrala och standardmässiga mått på genetisk variation, så kallade EVB:er - Essentiella Biodiversitets-Variabler, och har använts i Sverige sedan 2020.
Effektiv populationsstorlek (Ne)
mäter den genetiskt effektiva storleken hos populationer. Det är en standardparameter inom populationsgenetik som kopplar till en populations förmåga att utveckla nya anpassningar (adaptiv förmåga). Riktvärde för en god adaptiv potential är Ne ≥ 500 och detta är också en huvudindikator inom ramen för FN:s konvention om biologisk mångfald. I Sverige används fler tröskelvärden där Ne < 50 leder till röd varning p.g.a. mycket låg adaptiv förmåga.
Genetisk mångfald inom populationer (ΔH)
mäter skillnader i genetisk variation inom population över tid. Beroende på vilka markörer som undersöks används olika EVB:er för genetisk variation för att beräkna tillståndsvärdet:
- Förväntad och observerad heterozygozitet (HE; HO)
- Antal alleler per loci (NA)
- Allelic richness (AR)
- Proportion polymorfa loci (PL)
- Nukleotiddiversitet (π)
- Inavelsgrad (FROH)
Genetisk mångfald mellan populationer (ΔFST)
mäter i vilken utsträckning genetiskt särpräglade populationer (vanligen lokalt anpassade populationer) bibehålls över tid. Den mäter även i vilken utsträckning graden av genetisk skillnad mellan bestånd bibehålls och därmed om det genetiska utbytet (konnektiviteten) förändras över tid.
För att tolka statistiskt säkerställda förändringar i mätvariabler (till exempel EBV:er) som ingår i tillståndsvärdena tillämpas tröskelvärden. Dessa bygger i sin tur på vetenskapligt föreslagna riktlinjer. Mer information finns i Andersson et al. (2022) samt i övervakningsmanualerna för respektive art.
Provtagningen sker genom fastlagda rutiner i enlighet med artspecifika övervakningsmanualer.
I avsaknad av nationellt datavärdskap kommer både mätdata och de beräknade indikatorerna att lagras hos Havs- och vattenmyndigheten, HaV, och göras fritt tillgängliga för nedladdning. För närvarande pågår utvecklingen av formatet för dessa primära mätdata. När formatet har fastställts kommer information om hur och var dessa data kan laddas ner att publiceras.
Då genetisk inomartsvariation är ett förhållandevis nytt övervakningsprogram och forskning på berörda arter pågår parallellt. Därför sker kontinuerlig utveckling. Datavärdskap och övervakningsmanualer håller på att tas fram.
Programmet avser också utökas till flera arter. Exempel på prioriterade arter är abborre, blåmussla, skarpsill, tånglake och vikaresäl.
Vad styr vad som ska övervakas och hur används data?
Vad Sverige ska övervaka styrs av en rad nationella och internationella lagstiftningar, rapporteringskrav och konventioner. Dessa styr också hur övervakningsdata ska användas för att följa upp och bedöma statusen i den akvatiska miljön.
Om det saknas metoder för att bedöma status baserat på övervakningsdata pågår oftast ett utvecklingsarbete i internationella expertgrupper där svenska experter deltar. Att bedömningsmetoder saknas kan ibland bero på brister i data och i andra fall på svårigheter att fastställa gränser för bedömning av status.
Vattendirektivet fokuserar bland annat på miljökvalitetsnormer i vattenmiljö vilket inkluderar god konnektivitet. Övervakning av genetisk variation ger en god bild av biologisk konnektivitet i vattenmiljöer och kan därför användas i rapportering och i förvaltning.
Havsmiljödirektivet fokuserar på en god miljöstatus i havsmiljö och där är genetisk mångfald en nyckelfaktor för populationers och arters ekosystemfunktion och överlevnadsförmåga liksom för genetiskt utbyte mellan skyddade områden.
Artikel 17 om Art- och habitatdirektivet där information från genetisk övervakning visar på i vilken utsträckning potential för långsiktig överlevnad och anpassningsförmåga föreligger.
Att genomföra Naturrestaureringsförordningen kan stödjas av övervakning av genetisk mångfald. Den genetiska informationen kommer att kunna användas för flera av de mål som förordningen utformat.
Konventionen om biologisk mångfald har ett nytt globalt ramverk för biodiversitet sedan 2022 som sätter stort fokus på genetisk mångfald. Övergripande tillståndsmål för 2050 är till exempel att populationer av arter ska vara tillräckligt stora för att bibehålla genetisk anpassningsförmåga (Ne>500).
Ett av aktionsmålen till 2030 är att genetisk mångfald både inom och mellan populationer av arter ska bibehållas och anpassningsförmåga säkerställas. En huvudindikator som sammanfaller med en av HaV:s indikatorer (Ne>500) ska rapporteras av länderna från 2026. En kompletterande indikator som överensstämmer med HaV:s ΔFST-indikator finns också för att stödja måluppfyllelse.
Data nyttjas i Sveriges CBD-rapportering gällande både huvudindikatorn A.4 Ne och komplementindikatorn A.CY.22 Populations maintained. Både proxydata och DNA-data används.
Havsplanering genomförs i Sverige utifrån kraven i havsplaneringsförordningen och är till för att visa hur havet utanför kustzonen ska användas effektivt och hållbart, nu och i framtiden.
I havsplaneringen görs en miljöbedömning för att studera havsplanernas konsekvenser för havsmiljön. Det är en del i tillämpningen av ekosystemansatsen. Syftet med miljöbedömningen är bland annat att integrera miljöaspekter i havsplanerna för att främja en hållbar utveckling. HaV har utvecklat ett planeringsverktyg som heter Symphony för att rumsligt uppskatta miljöpåverkan och känslighet i ekosystemen. Övervakningen av genetisk inomartsvariation är relevant för havsplaneringen och data kan ingå i underlag för konsekvensbedömning i havsplaneringen.
Svenska miljökvalitetsmålen
För att följa upp de svenska miljökvalitetsmålen behövs dataunderlag. Övervakning av genetisk inomartsvariation ger underlag till preciseringarna om god miljöstatus och gynnsam bevarandestatus och genetisk variation i
De svenska miljömålen följs upp genom en årlig uppföljning på nationell och regional nivå. Det görs även en fördjupad utvärdering vart fjärde år.
Mer information om miljökvalitetsmålen
Agenda 2030
Av FN:s 17 globala miljömål kan data på genetisk inomartsvariation användas för att följa upp
mål 14 – Hav och marina resurser, delmålet 14.2
"Senast 2020 förvalta och skydda marina och kustnära ekosystem på ett hållbart sätt för att undvika betydande negativa konsekvenser, bland annat genom att stärka deras motståndskraft, samt vidta åtgärder för att återställa dem i syfte att uppnå friska och produktiva hav."
mål 15 – Ekosystem och biologisk mångfald, delmålet 15.5
"Vidta omedelbara och betydande åtgärder för att minska förstörelsen av naturliga livsmiljöer, hejda förlusten av biologisk mångfald och senast 2020 skydda och förebygga utrotning av hotade arter."



